Würzburg. Stellen Sie sich eine zerknitterte Zeitung vor. Im dreidimensionalen Raum nimmt sie viel mehr Platz ein, aber die gleichen Informationen und Nachrichten befinden sich immer noch auf einer niederdimensionalen Struktur, der Zeitung selbst. Um besser lesen zu können, muss die flache Form der Zeitung wiederhergestellt werden. Ähnliches macht MARBLE mit den neuronalen Aktivitätsmustern im Gehirn. Die KI-Methode reduziert diese komplexen, hochdimensionalen Datensätze auf einfache Strukturen, so genannte Mannigfaltigkeiten. MARBLE steht für MAnifold Representational Basic Learning.
Robert Peach, Physiker und Computational Neuroscientist in der Neurologischen Klinik des Universitätsklinikums Würzburg (UKW), und Adam Gosztolai, Mathematiker an der Medizinischen Universität Wien, entwickelten MARBLE gemeinsam mit ehemaligen Kollegen und Vorgesetzten vom Imperial College in London und der École Polytechnique Fédérale de Lausanne (EPFL) in der Schweiz. Ihre technische Innovation, die das alltägliche Leben auf vielfältige Weise verbessern könnte, stellen Gosztolai und Peach als Erstautoren in der renommierten Fachzeitschrift Nature Methods vor.
MARBLE erkennt und interpretiert neuronale Hirnaktivitäten
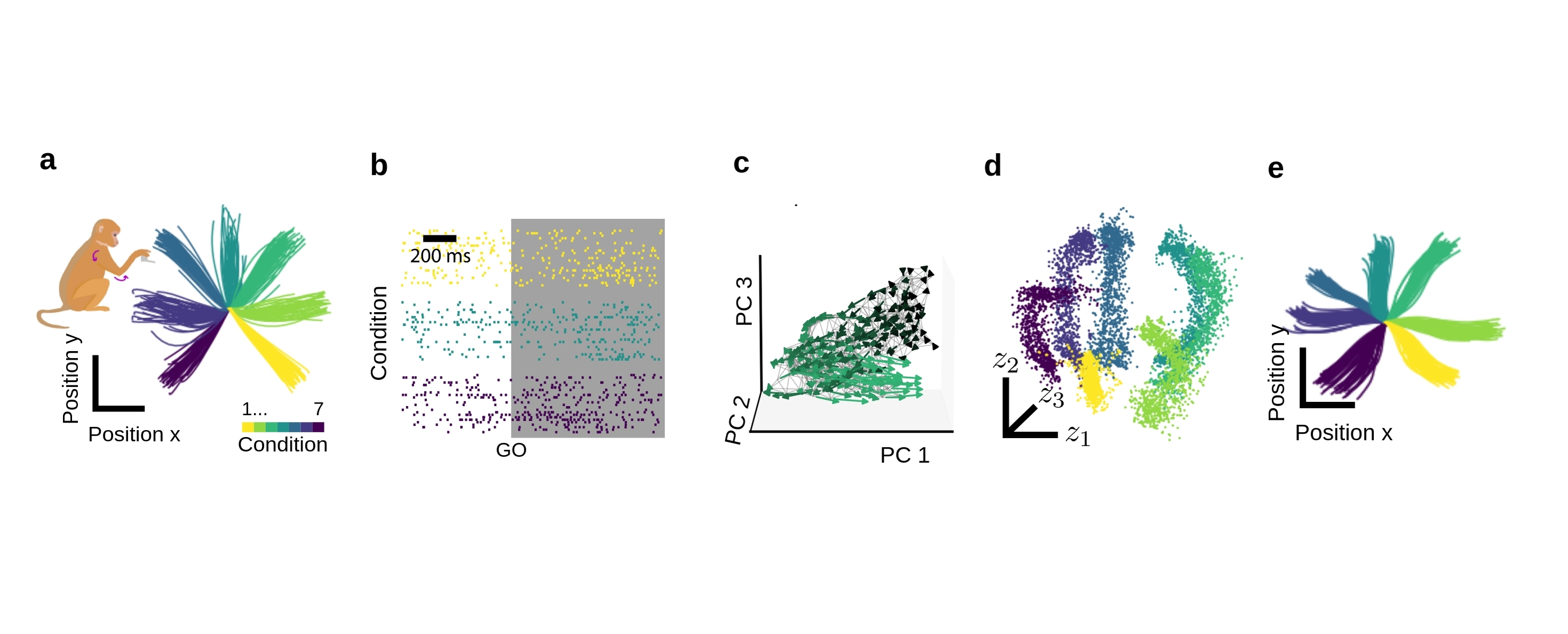
Hinter der Entwicklung des computergestützten Werkzeugs MARBLE steht eine zentrale Frage: Verwenden zwei Gehirne bei der Lösung ähnlicher Aufgaben gleiche oder unterschiedliche Denkstrategien? Statt alle Neuronen einzeln zu untersuchen, betrachtet MARBLE nur Ausschnitte der Aktivität und vergleicht sie zwischen verschiedenen Spezies und Aufgaben. Dazu zerlegt MARBLE die neuronalen Signale in charakteristische Aktivitätsmuster, die Robert Peach „Puzzleteile“ nennt. Um mit den geschwungenen Strukturen umzugehen, die bei komplexen, nichtlinearen Hirnprozessen häufig auftreten, verwenden die Wissenschaftler ein spezialisiertes geometrisches Deep-Learning-Netzwerk, das die Puzzleteile in ihrer Dynamik, also ihrer Bewegung in Raum und Zeit, erkennt und in eine verständliche Form bringt.
Gemeinsamkeiten zwischen verschiedenen Individuen finden, ohne sie in ein starres Schema zu pressen
Die Forscher testeten MARBLE an künstlichen neuronalen Netzen, simulierten Systemen und echten Hirndaten von Primaten und Nagetieren. Dabei fanden sie wiederkehrende Muster, die mit Denkprozessen wie Entscheidungsfindung oder Anpassung an neue Situationen zusammenhängen. „Das heißt, wenn verschiedene Tiere die gleiche Strategie anwenden, teilen sie sich diese Puzzleteile, betten sie aber in ihre eigene, individuell gekrümmte Struktur ein“, erklärt Robert Peach. Und das sei der entscheidende Vorteil gegenüber bisherigen Methoden. MARBLE kann eine gemeinsame Struktur im Denken erkennen, ohne die einzigartige „Sprache“ jedes Gehirns zu ignorieren.
Während herkömmliche Methoden oft nur statische Muster betrachten oder Daten über viele Experimente hinweg mitteln, erkennt MARBLE zeitliche Veränderungen in den Signalen und kann so feine Unterschiede zwischen den Denkstrategien erkennen. Peach: „Unser Ansatz arbeitet mit nur wenigen Vorgaben von außen und ohne feste Verhaltensregeln, so dass die Analyse objektiver bleibt.“
Präzisere Steuerung von Prothesen und anderen Hilfsmitteln
Vor allem Menschen mit motorischen Einschränkungen könnten von dieser technischen Innovation profitieren. Denn wenn man besser versteht, wie das Gehirn im Laufe der Zeit arbeitet, lassen sich fortschrittlichere Gehirn-Computer-Schnittstellen entwickeln, die eine präzisere Steuerung von Prothesen und anderen Hilfsmitteln ermöglichen. Dieses Ziel verfolgt unter anderem der von der Deutschen Forschungsgemeinschaft (DFG) geförderte transregionale Sonderforschungsbereich (SFB) TRR 295 ReTune, in dem sich das UKW gemeinsam mit der Charité - Universitätsmedizin Berlin mit spezifischen Aspekten von Störungen motorischer Netzwerke beschäftigt. Daher wurde auch die Forschung von Robert Peach im Rahmen von ReTune gefördert. Prof. Dr. Jens Volkmann, Direktor der Klinik und Poliklinik für Neurologie am UKW, ist stellvertretender Sprecher des TRR, der im vergangenen Sommer in die zweite Förderphase ging.
Fortschritte in Gesundheitsversorgung, Barrierefreiheit und Mensch-Computer-Interaktion
Darüber hinaus hilft das Forschungsprojekt, besser zu verstehen, wie das gesunde Gehirn Aufmerksamkeit steuert und Neues lernt. Diese Erkenntnisse könnten neue Ansätze für die kognitive Leistungssteigerung oder die Rehabilitation nach Schlaganfällen inspirieren. Selbst alltägliche Technologien – wie digitale Assistenten oder tragbare Geräte – könnten von Algorithmen profitieren, die sich daran orientieren, wie das Gehirn komplexe Aufgaben in Echtzeit bewältigt. Robert Peach fasst zusammen: „Wenn wir lernen, die verborgenen Muster hinter neuronalen Prozessen zu entschlüsseln, können wir Werkzeuge entwickeln, die natürlicher mit unserem Geist und Körper interagieren – mit möglichen Fortschritten in Gesundheitsversorgung, Barrierefreiheit und der Mensch-Computer-Interaktion.“
Im nächsten Schritt will das Team MARBLE auf komplexere Datensätze und verschiedene Spezies anwenden und eng mit klinischen Partnern zusammenarbeiten, um das Potenzial für die Behandlung von Bewegungsstörungen zu erforschen. Außerdem sollen die zugrundeliegenden mathematischen Methoden weiterentwickelt und verfeinert werden, um genauere Einblicke in die dynamischen Prozesse des Gehirns zu gewinnen.
Das Forschungsprojekt wurde gefördert von der DFG im Rahmen von ReTune sowie vom Engineering and Physical Sciences Research Council (EPSRC), dem Human Frontiers Science Programme und dem schweizerischen Blue Brain Project.
Text: KL / Wissenschaftskommunikation
Publikation:
Gosztolai, A., Peach, R.L., Arnaudon, A. et al. MARBLE: interpretable representations of neural population dynamics using geometric deep learning. Nat Methods (2025). https://doi.org/10.1038/s41592-024-02582-2
Research Briefing: www.nature.com/articles/s41592-024-02581-3